Bioinformática - Análise Filogenética com Clustalx
O Clustalx é um software para o alinhamento de sequências nucleotídicas, peptídicas e analises filogênicas. O Clustalx é software semi-livre segundo a sua forma de licenciamento. Embora possua características de software livre como permissão de execução de programa, não há permissão de uso para fins comerciais.
[ Hits: 28.323 ]
Por: José Cleydson Ferreira da Silva em 08/07/2010
Usando o Clustalx

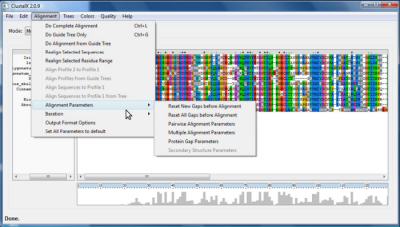
- Gap Opening- penalização de escore para iniciar uma região de "Gap".
- Gap Extension- penalização de escore para estender uma região de "Gap" (normalmente menor que o Gap Opening)
- Delay Divergent Sequences - atraso de alinhamento de sequências divergentes que somente serão alinhadas após as outras sequências (porcentagem de identidade abaixo da qual a sequência será considerada divergente)
- Transition Weight (somente DNA) - da a transições (A<->G, T<->C) um escore diferente de 0.
- Use negative matrix - permite o uso de matrizes negativas, importante quando as sequências forem relacionadas somente em uma pequena porção. Em condições normais prejudica um pouco o alinhamento.
- Protein Weight Matrix - matriz de substituição a ser utilizada. Note que você só escolhe a serie de matriz: Blosum, PAM etc, o tipo de matriz dentro desta série (por exemplo Blosum 62, blosum 80) é escolhido automaticamente pelo programa.
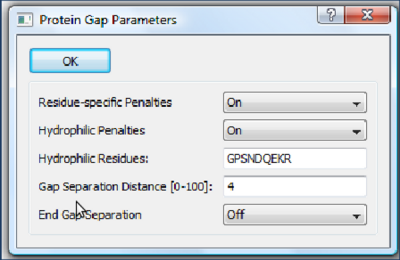
- Residue-specific Penalties - considera a vizinhança de alguns resíduos como mais ou menos favorável para a abertura de "Gaps".
- Hydrophilic Penalties - aumenta a chance de Gaps em regiões ricas em resíduos hidrofílicos que, usualmente, representam regiões menos estruturadas.
- Hydrophilic Residues - especifica quais resíduos são considerados hidrofílicos.
- Gap Separation Distance - número de resíduos de distância de uma região com Gap na qual é penalizada uma nova abertura de Gap.
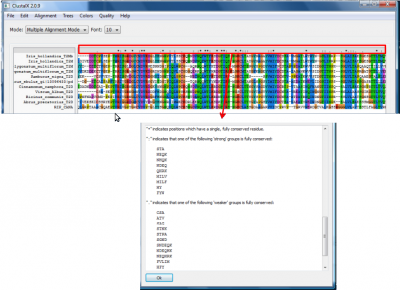
A apresentação de resultado de um alinhamento no ClustalX é representada por símbolos que indicam a conservação e os resíduos ou grupos de resíduos em uma coluna.
- Low Scoring Segments - mostra, em cinza, regiões com baixo escore e que, portanto, não seriam muito confiáveis
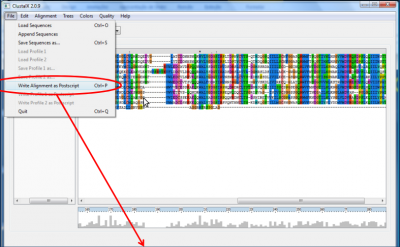
Também é possível exportar o alinhamento em formato Postscript através dos atalhos Ctrl+P ou acionando o menu File opção Write Alignment as Postscript. A visualização do arquivo pode ser feita por meio de um visualizador de imagens.
2. Usando o Clustalx
3. Sobre o autor
Bioinformática - PhyML: alinhamento de sequências nucleotídicas em ambiente paralelo
Como migrar banco de dados MySQL para PostgreSQL
Conheça tudo sobre os hardwares que compõem o seu computador com um simples comando
Cairo-Dock - Seu desktop Linux com cara de MAC
Bioinformática - Clustalw-MPI: Análise Filogenética utilizando computação paralela e distribuída
Instalação e correção de erros do DBDesigner 4 no Linux
Customizando e atualizando o Slax
Otimizando o Sabayon Linux (inclusive o Firefox)
Isoqlog no Debian - Analisando os logs do seu MTA
Implementando um Linux Terminal Service (LTS) no Debian
Nenhum comentário foi encontrado.
Patrocínio
Destaques
Artigos
Programa IRPF - Guia de Instalação e Resolução de alguns Problemas
Criando uma Infraestrutura para uma micro Empresa
Criar entrada (menuentry) ISO no Grub
Como gerar qualquer emoji ou símbolo unicode a partir do seu teclado
Dicas
Instalando o Pi-Hole versão v5.18.4 depois do lançamento da versão v6.0
Instalar o VIM 9.1 no Debian 12
Como saber o range de um IP público?
Muitas dificuldades ao instalar distro Linux em Notebook Sony Vaio PCG-6131L (VPCEA24FM)
Tópicos
Habilitar a opção de autocadastro no Ocomon 6.0.2 (2)
Erro ao instalar o ChatGPT Desktop (8)
Liberação de alguns links no squid (7)
Instalação do Kraiyin-CRM em Linux dentro de VMs no VirtualBox (1)
Top 10 do mês
-

Xerxes
1° lugar - 81.607 pts -

Fábio Berbert de Paula
2° lugar - 60.076 pts -

Buckminster
3° lugar - 23.656 pts -

Mauricio Ferrari
4° lugar - 19.812 pts -
Alberto Federman Neto.
5° lugar - 18.043 pts -

Daniel Lara Souza
6° lugar - 16.043 pts -

Diego Mendes Rodrigues
7° lugar - 15.687 pts -

edps
8° lugar - 15.679 pts -

Alessandro de Oliveira Faria (A.K.A. CABELO)
9° lugar - 15.192 pts -

Andre (pinduvoz)
10° lugar - 13.189 pts