Bioinformática - Análise Filogenética com Clustalx
O Clustalx é um software para o alinhamento de sequências nucleotídicas, peptídicas e analises filogênicas. O Clustalx é software semi-livre segundo a sua forma de licenciamento. Embora possua características de software livre como permissão de execução de programa, não há permissão de uso para fins comerciais.
[ Hits: 28.316 ]
Por: José Cleydson Ferreira da Silva em 08/07/2010
Instalação do Clustalx
Sobre o Clustalx
O Clustalx é um software para o alinhamento de sequências nucleotídicas, peptídicas e analises filogênicas. O Clustalx é software semi-livre segundo a sua forma de licenciamento. Embora possua características de software livre como permissão de execução de programa, de estudo de seu funcionamento, de liberdade de redistribuição e de modificação não há permissão de uso para fins comerciais.O código fonte pode ser obtido no site do projeto. O processo de instalação pode ser feito de duas formas: por meio do código fonte ou pelo gerenciador de pacotes das distribuições Linux derivadas do Debian. Porém é necessário possuir repositórios non-free configurados no arquivo /etc/apt/sources.list.
Instalação do Clustalx
A instalação feita por meio de gerenciadores de pacotes em derivados do Debian, como o Ubuntu por exemplo, pode ser feita com o seguinte comando.# apt-get install clustalx
Caso opte pela instalação por meio do código fonte, os seguintes comandos fazem o download e, em seguida, a compilação.
Fazer download do fonte:
wget http://www.clustal.org/download/current/clustalx-2.0.12.tar.gz
Compilar o fonte:
qmake
# make
Acessando o software
O acesso ao software pode se dar pelo uso do terminal por meio do comando:# clustalx &
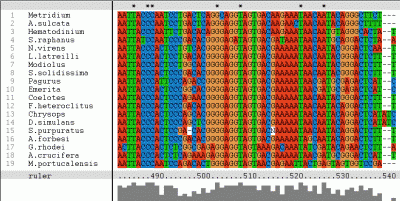
Ou, se optar por acessá-lo em modo "gráfico", use caminho através do menu Aplicativos => Ciência => Clustalx. A interface do software possui poucos menus; por esse motivo, a maior parte da tela é reservada para a análise das sequências.
O alinhamento de múltiplas sequências se dá:
- Pelo estabelecimento de relações filogenéticas entre diferentes sequências;
- Pela predizer de domínios conservados;
- Pela comparação da proteína desejada de forma ampla com os membros da mesma família da proteína.
2. Usando o Clustalx
3. Sobre o autor
Compiz - Janelas à 360 graus no Linux
Implementando rotas estáticas no Linux com route
Elaborando vídeo-aula no Linux com Gtk-recordMydesktop
Bioinformática - Clustalw-MPI: Análise Filogenética utilizando computação paralela e distribuída
Gerência de projetos com Redmine
Softwares para Biólogos no Linux
Planejando a migração de aplicações comerciais para o Linux/GNU
Implantando o Cacti (gerador de gráficos) em HTTP Seguro (HTTPS)
VirtualBox com PHPvirtualbox em Servidor Ubuntu 14.04
Nenhum comentário foi encontrado.
Patrocínio
Destaques
Artigos
Programa IRPF - Guia de Instalação e Resolução de alguns Problemas
Criando uma Infraestrutura para uma micro Empresa
Criar entrada (menuentry) ISO no Grub
Como gerar qualquer emoji ou símbolo unicode a partir do seu teclado
Dicas
Instalando o Pi-Hole versão v5.18.4 depois do lançamento da versão v6.0
Instalar o VIM 9.1 no Debian 12
Como saber o range de um IP público?
Muitas dificuldades ao instalar distro Linux em Notebook Sony Vaio PCG-6131L (VPCEA24FM)
Tópicos
Atualização do Google Chrome do Ubuntu (1)
Jogos baixados na Central de Aplicativos mas que não abrem (0)
Top 10 do mês
-

Xerxes
1° lugar - 80.579 pts -

Fábio Berbert de Paula
2° lugar - 59.480 pts -

Buckminster
3° lugar - 23.542 pts -

Mauricio Ferrari
4° lugar - 19.372 pts -
Alberto Federman Neto.
5° lugar - 17.855 pts -

Diego Mendes Rodrigues
6° lugar - 15.549 pts -

Daniel Lara Souza
7° lugar - 15.504 pts -

edps
8° lugar - 15.436 pts -

Alessandro de Oliveira Faria (A.K.A. CABELO)
9° lugar - 14.910 pts -

Andre (pinduvoz)
10° lugar - 12.701 pts