Montagem de Cluster
Artigo referente à montagem completa de um Cluster com o sistema Debian Wheezy, podendo ser adaptado para outras distribuições.
[ Hits: 146.145 ]
Por: Buckminster em 08/11/2013
Cluster 5
MPI (Message Passing Interface), em uma tradução livre, Interface de Passagem de Mensagens. É uma biblioteca com funções para troca de mensagens que faz a comunicação e a sincronização de processos em um Cluster paralelo.
Essa é a biblioteca que fará todo o trabalho de processamento no Cluster. Essa é a biblioteca que transforma um conjunto de máquinas em um Cluster.
Os objetivos do MPICH é fornecer uma implementação MPI que suporte eficientemente, diferentes plataformas de computação e comunicação, incluindo grupos de commodities (sistemas de Desktop, sistemas de memória compartilhada e arquiteturas multicore), redes de alta velocidade e sistemas de computação High-end de propriedade (Blue Gene, Cray); e permitir a pesquisa de ponta em MPI através de uma estrutura modular fácil de estender para outras implementações derivadas.
Servidor e nós:
# cd /usr/src
# wget http://www.mpich.org/static/downloads/3.0.4/mpich-3.0.4.tar.gz
# tar -xzvf mpich-3.0.4.tar.gz
# ls
# cd mpich-3.0.4
#./configure --help | less
# ./configure --prefix=/opt/mpich --enable-shared --enable-f95 --enable-threads=runtime --enable-romio --enable-nemesis-shm-collectives --enable-debuginfo CC=gcc CXX=g++ FC=gfortran F77=gfortran --with-pvfs2=/opt/pvfs2 --with-file-system="pvfs2+nfs" --with-thread-package=posix --with-device=ch3:nemesis --with-java=/etc/java-7-openjdk # Somente se for trabalhar com Java
Se não tiver erros, terminará com:
Configuration completed
# make
O make deverá terminar com a frase abaixo sem nenhuma mensagem de erro antes dela:
make[1]: Saindo do diretório '/usr/src/mpich-3.0.4'
# make install
O make install, idem ao make, não deverá apresentar erros.
Configurando as variáveis:
# vim ~/.bashrc
export PATH
E acrescentar, também, o caminho na variável LD_LIBRARY_PATH:
O arquivo bashrc acima com as variáveis, é o mesmo para o servidor e os nós.
Reinicie:
# shutdown -r now
Vendo as informações:
# mpiexec -info
Vamos testar nossa instalação, compilando e executando:
# cd /usr/src/mpich-3.0.4/examples
# ls
# mpicc -o cpi cpi.c # Esse arquivo deve ser compilado em todas as máquinas; acesse uma por uma ou utilize o seguinte comando:
# mpicc -o -hostfile /opt/hostfile /usr/src/mpich-3.0.4/examples/cpi.c cpi
O arquivo cpi.c realiza cálculo do PI.
Testando:
# mpirun -hostfile /opt/hostfile -n 7 /usr/src/mpich-3.0.4/examples/cpi
Para ver o manual, execute:
# man mpicc
Ou:
# man mpi_Barrier
Ou:
# man mpiexec
Para remover, em caso de erros e começar de novo:
# cd /usr/src/mpich-3.0.4
# make uninstall
# make distclean
# cd /usr/src/pvfs-2.8.2
# make distclean
E remova os seguintes arquivos com rm -rf:
- /usr/src/mpich-3.0.4
- /usr/src/pvfs-2.8.2
- /opt/mpich
- /opt/pvfs2
- /usr/local/sbin/pvfs2-server
Reinicie a máquina e recomece do princípio, as duas instalações criando novamente os diretórios /opt/mpich e /opt/pvfs2.
12. Ganglia
O Ganglia é um sistema de monitoramento distribuído escalável para sistemas de computação de alto desempenho, como Clusters e grids. Ele é baseado em um projeto hierárquico dirigido a Clusters. Ele aproveita as tecnologias amplamente utilizadas, tais como XML para representação de dados, XDR para compacto, transporte de dados portátil e RRDtool para o armazenamento e visualização de dados.
Ele usa as estruturas de dados e algoritmos projetados cuidadosamente para alcançar baixos custos gerais por nós e alta concorrência. A implementação é robusta e foi portada para um amplo conjunto de sistemas operacionais e arquiteturas de processadores, e está atualmente em uso em milhares de grupos em todo o mundo.
Ele tem sido usado para ligar os Clusters através de campi universitários e em todo o mundo, e pode ser escalado para lidar com grupos com 2000 nós.
Nos nós:
# apt-get update
# apt-get install ganglia-monitor
No servidor:
# apt-get update
# apt-get install apache2
Testando o Apache: abra o navegador e digite: localhost, deverá aparecer: It works.
# apt-get install php5 libapache2-mod-php5
Crie o arquivo:
# vim /var/www/info.php
Coloque dentro dele:
phpinfo();
?>
Salve e saia. Reinicie o Apache:
# /etc/init.d/apache2 restart
Abra o navegador e digite localhost/info.php, deverá aparecer a página de informações do PHP.
Instalando o Ganglia:
# apt-get install ganglia-webfrontend ganglia-monitor
Irá pedir duas vezes para reiniciar o Apache 2, faça.
Vamos copiar o arquivo necessário:
# cp /etc/ganglia-webfrontend/apache.conf /etc/apache2/sites-enabled/ganglia.conf
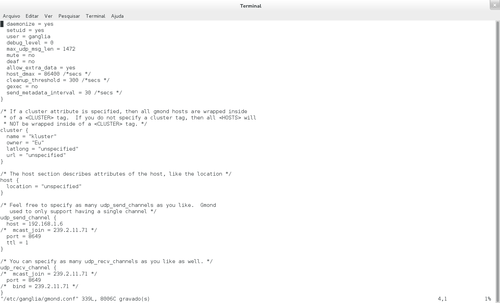
Alterar o arquivo /etc/ganglia/gmod.conf no servidor e copiar para os nós. É extremamente necessário, tendo em vista que a conexão remota fica muito lenta, devido ao IP 239.2.11.71.
A parte que nos interessa alterar, está na imagem acima. Comente as linhas onde tem o IP 239.2.11.71, ou similar, e configure o host com o IP do servidor.
Altere o arquivo /etc/ganglia/gmetad.conf no servidor, deixando como na imagem acima, não sendo necessário copiar para os nós.
Para reiniciar o serviço nos nós:
# service ganglia-monitor restart
Reiniciar o Apache e o Ganglia no servidor:
# /etc/init.d/apache2 restart
# service gmetad restart
A partir daí é só digitar no navegador localhost/ganglia.
2. Configurações de Hardware e Software
3. Cluster 1
4. Cluster 2
5. Cluster 3
6. Cluster 4
7. Cluster 5
8. Cluster 6
9. Conclusão
Instalar Minecraft, League of Legends e Fortnite no Linux
Instalar e configurar o Nftables com exemplos básicos de configurações
Instalando e Configurando o pgAgent no Linux (pgAdmin e PostgreSQL)
Manual do IPtables - Comentários e sugestões de regras
"TORIFICANDO" todo um sistema GNU/Linux utilizando a rede TOR
Tutorial de instalação e configuração do CACIC 3.1.15
Balanceamento de links - Load balance + Failover + Failback
[1] Comentário enviado por danniel-lara em 08/11/2013 - 08:06h:
Parabéns pelo Artigo
Obrigado tchê.
[4] Comentário enviado por micnet em 08/11/2013 - 12:22h:
Muito Bom Amigo! Parabéns!!
Obrigado.
[5] Comentário enviado por suxfuuu em 08/11/2013 - 13:41h:
Nossa show de bola parabéns!
Obrigado.
Material muito bom, e um excelente teste.
Pergunta: Você conseguiu fazer uma avaliação do consumo de energia com o processamento e em espera?
Seria interessante ter uma noção de quanto de energia está sendo consumida.
O legal também, é que o nobreak que você escolheu, se não me engano tem gerenciamento (horrível no linux) porém, um ponto de partida de avaliação de consumo.
Tenho um aqui do SMS, porém difícil de verificar com linux, mesmo com USB.
O aquecimento também é um baita problema com tantas máquinas... rs...
o Artigo é excelente, favoritado.
uso Modelagem Molecular na Faculdade
[8] Comentário enviado por adrianoh2 em 09/11/2013 - 19:45h:
Material muito bom, e um excelente teste.
Pergunta: Você conseguiu fazer uma avaliação do consumo de energia com o processamento e em espera?
Seria interessante ter uma noção de quanto de energia está sendo consumida.
O legal também, é que o nobreak que você escolheu, se não me engano tem gerenciamento (horrível no linux) porém, um ponto de partida de avaliação de consumo.
Tenho um aqui do SMS, porém difícil de verificar com linux, mesmo com USB.
O aquecimento também é um baita problema com tantas máquinas... rs...
Obrigado.
Ainda não fiz uma avaliação da energia consumida, mas pretendo fazer.
Os nobreaks não estão sendo gerenciados, por enquanto as conexões são somente físicas. Priorizei a autonomia com baterias externas.
Mas pretendo instalar um gerenciamento.
Não estou tendo problemas com aquecimento, a sala aqui é bastante ventilada e algumas máquinas tem ventoinhas extras. Como pode ver na foto, deixei um espaço entre as máquinas.
[9] Comentário enviado por MarcosLnx em 09/11/2013 - 23:55h:
Muito bom o material, parabéns...
Obrigado.
[10] Comentário enviado por albfneto em 10/11/2013 - 10:33h:
o Artigo é excelente, favoritado.
uso Modelagem Molecular na Faculdade
Obrigado Alberto.
Podemos trocar umas idéias sobre Modelagem Molecular. Faz pouco tempo que estou trabalhando com isso (um ano aproximadamente), mas estou me aprofundando nos estudos.
Resolvi postar esse artigo tendo em vista a falta de material prático em Língua Portuguesa sobre o assunto.
Não sou da área, mas tenho que postar meu elogio.
Artigo de alto nível.
[14] Comentário enviado por izaias em 10/11/2013 - 17:48h:
Não sou da área, mas tenho que postar meu elogio.
Artigo de alto nível.
Obrigado Izaias.
Esse material é raro...já havia feito varias pesquisas mas sem sucesso,e o que tinha era muito velho.
Agora os caras estão fazendo com PS3 por conta do processador.
http://g1.globo.com/Noticias/Tecnologia/0,,MUL146410-6174,00-UNICAMP+USA+PLAYSTATION+PARA+REALIZAR+P...
Valeuu"
[16] Comentário enviado por sayrus em 10/11/2013 - 21:01h:
Esse material é raro...já havia feito varias pesquisas mas sem sucesso,e o que tinha era muito velho.
Agora os caras estão fazendo com PS3 por conta do processador.
http://g1.globo.com/Noticias/Tecnologia/0,,MUL146410-6174,00-UNICAMP+USA+PLAYSTATION+PARA+REALIZAR+P....
Valeuu"
Obrigado.
Parabéns, ótimo artigo.
Favoritos.
[18] Comentário enviado por julio_hoffimann em 12/11/2013 - 00:08h:
Parabéns, ótimo artigo.
Favoritos.
Obrigado.
Fazendo uns testes com o Siesta, identifiquei que o desempenho dele no cluster utilizando o PVFS2 é bem maior do que com NFS.
Em duas simulações iguais, uma com o PVFS2 e outra com o NFS, a diferença foi de quase 50%, ou seja, o PVFS2 terminou o trabalho quase na metade do tempo do que com o NFS.
Acredito que seja porque o volume de dados que o Siesta calcula seja grande.
Desabilitei o NFS e deixei o PVFS2 funcionando.
Porém, continuo com os testes para ter certeza, pois ainda não é conclusivo.
OLá, boa tarde!
está de parabéns, bom eu vou fazer o projeto final um cluster , mas ainda estou estudando esse assunto, mas no momento preciso fazer um trabalho de avaliação para montar um cluster HPC
e configurar um pequeno cluster com Linux para execução de algoritmos paralelos (por ex. O caso do Laboratório de Metrologia de Nêutrons), se puder me passar algumas dicas por onde eu começo eu agradeço...
[21] Comentário enviado por silvafarol2 em 24/01/2014 - 15:38h:
OLá, boa tarde!
está de parabéns, bom eu vou fazer o projeto final um cluster , mas ainda estou estudando esse assunto, mas no momento preciso fazer um trabalho de avaliação para montar um cluster HPC
e configurar um pequeno cluster com Linux para execução de algoritmos paralelos (por ex. O caso do Laboratório de Metrologia de Nêutrons), se puder me passar algumas dicas por onde eu começo eu agradeço...
Comece planejando o cluster (quantas máquinas terão). Depois de escolhido o hardware (as máquinas), monte o cluster fisicamente e depois instale e configure os sistemas.
Somente depois faça a escolha das bibliotecas (MPI, Lapack, Blas, etc) e essas bibliotecas devem se adequar aos programas que você irá utilizar para a execução desses algoritmos.
A escolha do hardware de rede é importantíssima (placas de rede, switch, cabos, etc), devem ser de boa qualidade. O cabeamento deve ser todo ele no mesmo padrão.
Nas referências da página 'Conclusão' do artigo estão alguns links que você pode utilizar.
Mas veja bem, as bibliotecas a serem utilizadas dependem dos programas que você irá usar no cluster.
Eu utilizo a MPICH, mas de repente para você seja mais aconselhável utilizar a OpenMPI, ou outra. Isso depende dos programas que você irá utilizar.
Olá,
alguém pode mim ensinar como submeter um cálculo no siesta, por exemplo, quero rodar h2o, como devo fazer?
Buckminster,
Valew pelo artigo. Muito Boooom !!!
Gostei mais ainda por ter sido feito com o Debian.
Faz tempo que tava querendo algo um pouco mais explicado sobre o assunto e qdo tiver um pouco mais de tempo vou tentar a instalacao na pratica. Nos ultimos meses, nem sequer tenho tido tempo de sentar na frente do computador, quanto mais pensar em algo que vai consumir um bom tempo para realizar.
Vc por caso nao teria alguma referencia de leitura (preferencialmente disponivel na internet) para instalar os nodes sem os HDs (diskless) ?
Tava planejando algo com 6 a 12 Xeons L5420.
Bem... como eh algo que sempre quis tentar montar,
pode ser que demore um pouco... mas um dia concluo... rsss
[23] Comentário enviado por Damonff em 21/02/2014 - 23:00h:
Olá,
alguém pode mim ensinar como submeter um cálculo no siesta, por exemplo, quero rodar h2o, como devo fazer?
Você deverá ter os arquivos H.psf e O.psf e o arquivo h2o.fdf (<< esse arquivo .fdf você dá o nome que quiser).
Coloque esses arquivos dentro de uma pasta de sua escolha (crie uma pasta chamada h2o, por exemplo), sendo que esta pasta deverá ter somente os três arquivos citados.
Entre na pasta h2o e digite um comando similar a esse:
# /opt/siesta < h2o.fdf | tee h2o.out
onde /opt/siesta é o local onde você instalou o Siesta, caso você instalou em outro local mude /opt/siesta para o local certo.
No site abaixo você encontra os pseudopotenciais (pseudopotentials) do oxigênio e do hidrogênio, bem como todos os .psf da tabela periódica.
Escolha de acordo com o que você quer, LDA ou GGA:
http://departments.icmab.es/leem/siesta/Databases/Pseudopotentials/periodictable-intro.html
Aqui tem um exemplo de um h2o.fdf (é o mesmo que está em ~/siesta/Examples/H2O, mas é bastante básico:
https://github.com/id23cat/siesta-3.1/blob/master/Examples/H2O/h2o.fdf
[24] Comentário enviado por nqk18469 em 22/02/2014 - 09:21h:
Buckminster,
Valew pelo artigo. Muito Boooom !!!
Gostei mais ainda por ter sido feito com o Debian.
Faz tempo que tava querendo algo um pouco mais explicado sobre o assunto e qdo tiver um pouco mais de tempo vou tentar a instalacao na pratica. Nos ultimos meses, nem sequer tenho tido tempo de sentar na frente do computador, quanto mais pensar em algo que vai consumir um bom tempo para realizar.
Vc por caso nao teria alguma referencia de leitura (preferencialmente disponivel na internet) para instalar os nodes sem os HDs (diskless) ?
Tava planejando algo com 6 a 12 Xeons L5420.
Bem... como eh algo que sempre quis tentar montar,
pode ser que demore um pouco... mas um dia concluo... rsss
http://www.eelvex.net/administration/diskless-cluster-on-debian-installation-log/
http://jablonskis.org/2011/howto-to-build-a-diskless-linux-cluster/index.html
http://www.google.com.br/url?sa=t&rct=j&q=&esrc=s&source=web&cd=4&ved=0CFAQFjAD&url=http%3A%2F%2Fwww...
[26] Comentário enviado por Buckminster em 22/02/2014 - 12:01h:
http://www.eelvex.net/administration/diskless-cluster-on-debian-installation-log/
http://jablonskis.org/2011/howto-to-build-a-diskless-linux-cluster/index.html
http://www.google.com.br/url?sa=t&rct=j&q=&esrc=s&source=web&cd=4&ved=0CFAQF....
Muito obrigado pelos links.
Dei uma olhada por cima no conteudo dos 2 pimeiros links e ja baixei o .pdf para ir lendo no tablet.
Infelizmente estou de mudanca e meus atuais horarios no trabalho tambem nao ajudam e quaquer tentativa de montagem, principalmente no que tange ao cabeamente, teria que ser toda refeita.
Assim que possivel o inicio do projeto, apareco para dar informacoes sobre o andamento das coisas.
Abs.
Primeiramente meus parabéns por este excelente artigo!
- Vamos às dúvidas:
Na prática, como é feita a conexão física dos cabos?
Eu conecto o servidor num roteador e conecto os nós nesse roteador?
[28] Comentário enviado por rudregues em 31/03/2014 - 01:00h:
Primeiramente meus parabéns por este excelente artigo!
- Vamos às dúvidas:
Na prática, como é feita a conexão física dos cabos?
Eu conecto o servidor num roteador e conecto os nós nesse roteador?
Conecta o servidor e os nós em um switch, independentemente de internet ou não.
Se quiser internet no cluster veja a página 2 do artigo: "Foi conectado um cabo direto do modem/roteador à porta 1 do Switch para prover Internet ao cluster. Nas outras portas foram conectadas as máquinas (servidor e nós)."
Caso você queira mais segurança, pode conectar o servidor no roteador em uma placa de rede e a outra placa de rede do servidor em um switch para fazer a conexão com os nós. Nesse caso você deverá ter obrigatoriamente um firewall bem configurado no servidor, pois a internet passará por ele. Além do que, nesse caso teu servidor deverá ser uma máquina boa. Com essa configuração você poderá ter acesso externo ao cluster.
Certo. Outra dúvida:
- Vou construir o cluster para resolver uma implementação do algoritmo de Eliminação de Gauss. Então pelo que entendi o pacote "SIESTA" e o "Gromacs" não são necessários pra mim, certo?
- No passo "10. Servidor NFS"
[bold]# vim /etc/exports[/bold]
Coloque o conteúdo no final:
[code]/home/kluster 192.168.1.0/24(rw,all_squash,subtree_check,anonuid=150,anongid=100)
/opt 192.168.1.0/24(rw,all_squash,subtree_check)
/usr/local 192.168.1.0/24(rw,all_squash,subtree_check)[/code]
Deve ir o endereço da rede.
Salve e saia.
Veja bem, as pastas colocadas ali serão as pastas a serem compartilhadas pelo servidor com as outras máquinas do cluster. Então, todo e qualquer arquivo que deverá ser utilizado pelo cluster durante a execução de um programa deve ser colocado nesta mesma pasta.
1) O que você quis dizer com "Deve ir o endereço de rede"? Eu devo acrescentar outra coisa além?
2) E no caso dessas pastas que precisam conter os arquivos usados pelo cluster, o programa que vou usar ele pede um arquivo de texto de entrada com valores de uma matriz, nesse caso eu tenho que colocar esse arquivo nessas três pastas do servidor apenas ou em todos os nós também?
EDIT: 3) Para o meu objetivo, o Gromacs e o SIESTA não são necessários, tem algum outro que não seja essencial?
Se você for trabalhar somente com eliminação de Gauss, o SIESTA e o Gromacs não são necessários. Tem uma ou duas bibliotecas que não são necessárias também, mas instale todas como está no artigo, não irá prejudicar. Se futuramente você for usar algum programa de modelagem ou simulação computacional já estará pronto. O HPL também não é necessário, a não ser que você queria fazer testes de desempenho.
Você irá implementar um algoritmo específico de eliminação de Gauss em qual linguagem?
Ou você irá usar o Matlab?
Deve ir o endereço da rede, o exemplo do artigo está 192.168.1.0/24 (o endereço da rede geralmente termina em zero [0] seguido da máscara da rede [24]). Isso significa que as pastas serão compartilhadas por toda a rede do cluster.
As pastas ali colocadas são da sua escolha. Essas pastas é que serão compartilhadas entre si pelas máquinas do cluster.
E não precisa colocar o arquivo nas 3 pastas, colocando em uma é o suficiente.
Se quiser pode compartilhar somente uma pasta à sua escolha, daí o arquivo /etc/exports terá somente uma linha com um caminho de pasta e no /etc/fstab dos nós deverá acrescentar o mesmo caminho com a mesma pasta somente mudando o endereço de rede pelo IP do servidor como está no artigo.
A princípio, com o NFS ou o PVFS2 bem configurado você pode instalar os programas somente no servidor e colocar o arquivo da matriz somente no servidor.
Porém, dependendo do modo de execução do programa você deverá colocar nos nós também esse arquivo com a matriz.
Por exemplo, com o SIESTA eu tenho que colocar os arquivos de entrada em todas as máquinas (servidor e nós), porém, o programa está instalado somente no servidor.
Teste aí com o algoritmo e o arquivo de entrada com a matriz somente no servidor. Caso não der certo, coloque o arquivo nos nós também, tendo o cuidado de colocar com o mesmo nome e no mesmo caminho na mesma pasta em todas as máquinas.
Como último recurso, se não funcionar, instale o algoritmo em todas as máquinas (servidor e nós).
Obrigado por tirar essas dúvidas!
Então, o algoritmo é em C mesmo. Peguei dois algoritmos prontos na internet. O primeiro não lembro o link, mas o código fonte está aí:
/*
********************************************************************
Example 26 (gauss_elimination.c)
Objective : To solve the system of Linear Equations
using Gaussian Elimination without
pivoting on 'p' processors.
Input : Read files (mdatgaus.inp) for Matrix A
and (vdatgaus.inp) for Vector b
Output : The solution of matrix system of linear
equations Ax=b on processor 0.
Description : Input matrix is stored in n by n format.
Columnwise cyclic distribution of input
matrix is used for partitioning of the
matrix.
Necessary conditions : Number of Processes should be less than
or equal to 8.Matrix size for Matrix A
and vector size for vector b should be
equally striped, that is Matrix size and
Vector Size should be divisible by Number
of processes used.
********************************************************************
*/
#include <stdio.h>
#include <assert.h>
#include <mpi.h>
main(int argc, char** argv) {
/* .......Variables Initialisation ......*/
MPI_Status status;
int n_size, NoofRows_Bloc, NoofRows, NoofCols;
int Numprocs, MyRank, Root = 0;
int irow, jrow, icol, index, ColofPivot, neigh_proc;
double **Matrix_A, *Input_A, *Input_B, *ARecv, *BRecv;
double *Output, Pivot;
double *X_buffer, *Y_buffer;
double *Buffer_Pivot, *Buffer_bksub;
double tmp;
FILE *fp;
/* ........MPI Initialisation .......*/
MPI_Init(&argc, &argv);
MPI_Comm_rank(MPI_COMM_WORLD, &MyRank);
MPI_Comm_size(MPI_COMM_WORLD, &Numprocs);
/* .......Read the Input file ......*/
if(MyRank == 0) {
if ((fp = fopen ("./data/mdatgaus.inp", "r")) == NULL) {
printf("Can't open input matrix file");
exit(-1);
}
fscanf(fp, "%d %d", &NoofRows,&NoofCols);
n_size=NoofRows;
/* ...Allocate memory and read data .....*/
Matrix_A = (double **) malloc(n_size*sizeof(double *));
for(irow = 0; irow < n_size; irow++){
Matrix_A[irow] = (double *) malloc(n_size * sizeof(double));
for(icol = 0; icol < n_size; icol++)
fscanf(fp, "%lf", &Matrix_A[irow][icol]);
}
fclose(fp);
if ((fp = fopen ("./data/vdatgaus.inp", "r")) == NULL){
printf("Can't open input vector file");
exit(-1);
}
fscanf(fp, "%d", &NoofRows);
n_size=NoofRows;
Input_B = (double *)malloc(n_size*sizeof(double));
for (irow = 0; irow<n_size; irow++)
fscanf(fp, "%lf",&Input_B[irow]);
fclose(fp);
/* ...Convert Matrix_A into 1-D array Input_A ......*/
Input_A = (double *)malloc(n_size*n_size*sizeof(double));
index = 0;
for(irow=0; irow<n_size; irow++)
for(icol=0; icol<n_size; icol++)
Input_A[index++] = Matrix_A[irow][icol];
}
MPI_Bcast(&NoofRows, 1, MPI_INT, Root, MPI_COMM_WORLD);
MPI_Bcast(&NoofCols, 1, MPI_INT, Root, MPI_COMM_WORLD);
if(NoofRows != NoofCols) {
MPI_Finalize();
if(MyRank == 0){
printf("Input Matrix Should Be Square Matrix ..... \n");
}
exit(-1);
}
/* .... Broad cast the size of the matrix to all ....*/
MPI_Bcast(&n_size, 1, MPI_INT, 0, MPI_COMM_WORLD);
if(n_size % Numprocs != 0) {
MPI_Finalize();
if(MyRank == 0){
printf("Matrix Can not be Striped Evenly ....No of processors (p) should divide the size of the Input matrix (n)\n");
}
exit(-1);
}
NoofRows_Bloc = n_size/Numprocs;
/*......Memory of input matrix and vector on each process .....*/
ARecv = (double *) malloc (NoofRows_Bloc * n_size* sizeof(double));
BRecv = (double *) malloc (NoofRows_Bloc * sizeof(double));
/*......Scatter the Input Data to all process ......*/
MPI_Scatter (Input_A, NoofRows_Bloc * n_size, MPI_DOUBLE, ARecv, NoofRows_Bloc * n_size,
MPI_DOUBLE, 0, MPI_COMM_WORLD);
MPI_Scatter (Input_B, NoofRows_Bloc, MPI_DOUBLE, BRecv, NoofRows_Bloc, MPI_DOUBLE, 0,
MPI_COMM_WORLD);
/* ....Memory allocation of ray Buffer_Pivot .....*/
Buffer_Pivot = (double *) malloc ((NoofRows_Bloc + 1 + n_size * NoofRows_Bloc) * sizeof(double));
/* Receive data from all processors (i=0 to k-1) above my processor (k).... */
for(neigh_proc = 0; neigh_proc < MyRank; neigh_proc++) {
MPI_Recv(Buffer_Pivot, NoofRows_Bloc * n_size + 1 + NoofRows_Bloc, MPI_DOUBLE, neigh_proc,
neigh_proc, MPI_COMM_WORLD, &status);
for(irow = 0; irow < NoofRows_Bloc; irow++){
/* .... Buffer_Pivot[0] : locate the rank of the processor received */
/* .... Index is used to reduce the matrix to traingular matrix */
/* .... Buffer_Pivot[0] is used to determine the starting value of
pivot in each row of the matrix, on each processor */
ColofPivot = ((int) Buffer_Pivot[0]) * NoofRows_Bloc + irow;
for (jrow = 0; jrow < NoofRows_Bloc; jrow++){
index = jrow*n_size;
tmp = ARecv[index + ColofPivot];
for (icol = ColofPivot; icol < n_size; icol++)
ARecv[index + icol] -= tmp * Buffer_Pivot[irow*n_size+icol+1+NoofRows_Bloc];
BRecv[jrow] -= tmp * Buffer_Pivot[1+irow];
ARecv[index + ColofPivot] = 0.0;
}
}
}
/* ......Buffer_bksuborary buffer allocation ....*/
Y_buffer = (double *) malloc (NoofRows_Bloc * sizeof(double));
/* ....Modification of row entries on each processor ...*/
/* ....Division by pivot value and modification ...*/
for(irow = 0; irow < NoofRows_Bloc; irow++){
ColofPivot = MyRank * NoofRows_Bloc + irow;
index = irow*n_size;
Pivot = ARecv[index + ColofPivot];
assert(Pivot!= 0);
for (icol = ColofPivot; icol < n_size; icol++){
ARecv[index + icol] = ARecv[index + icol]/Pivot;
Buffer_Pivot[index + icol + 1 + NoofRows_Bloc] = ARecv[index + icol];
}
Y_buffer[irow] = BRecv[irow] / Pivot;
Buffer_Pivot[irow+1] = Y_buffer[irow];
for (jrow = irow+1; jrow<NoofRows_Bloc; jrow++) {
tmp = ARecv[jrow*n_size + ColofPivot];
for (icol = ColofPivot+1; icol < n_size; icol++)
ARecv[jrow*n_size+icol] -= tmp * Buffer_Pivot[index + icol + 1 + NoofRows_Bloc];
BRecv[jrow] -= tmp * Y_buffer[irow];
ARecv[jrow*n_size+irow] = 0;
}
}
/*....Send data to all processors below the current processors */
for (neigh_proc = MyRank+1; neigh_proc < Numprocs; neigh_proc++) {
/* ...... Rank is stored in first location of Buffer_Pivot and
this is used in reduction to triangular form ....*/
Buffer_Pivot[0] = (double) MyRank;
MPI_Send(Buffer_Pivot, NoofRows_Bloc*n_size+1+NoofRows_Bloc, MPI_DOUBLE, neigh_proc,
MyRank, MPI_COMM_WORLD);
}
/*.... Back Substitution starts from here ........*/
/*.... Receive from all higher processors ......*/
Buffer_bksub = (double *) malloc (NoofRows_Bloc * 2 * sizeof(double));
X_buffer = (double *) malloc (NoofRows_Bloc * sizeof(double));
for (neigh_proc = MyRank+1; neigh_proc<Numprocs; ++neigh_proc) {
MPI_Recv(Buffer_bksub, 2*NoofRows_Bloc, MPI_DOUBLE, neigh_proc, neigh_proc,
MPI_COMM_WORLD,&status);
for (irow = NoofRows_Bloc-1; irow >= 0; irow--) {
for (icol = NoofRows_Bloc-1;icol >= 0; icol--) {
/* ... Pick up starting Index .....*/
index = (int) Buffer_bksub[icol];
Y_buffer[irow] -= Buffer_bksub[NoofRows_Bloc+icol] * ARecv[irow*n_size+index];
}
}
}
for (irow = NoofRows_Bloc-1; irow >= 0; irow--) {
index = MyRank*NoofRows_Bloc+irow;
Buffer_bksub[irow] = (double) index;
Buffer_bksub[NoofRows_Bloc+irow] = X_buffer[irow] = Y_buffer[irow];
for (jrow = irow-1; jrow >= 0; jrow--)
Y_buffer[jrow] -= X_buffer[irow] * ARecv[jrow*n_size + index];
}
/*.... Send to all lower processes...*/
for (neigh_proc = 0; neigh_proc < MyRank; neigh_proc++)
MPI_Send(Buffer_bksub, 2*NoofRows_Bloc, MPI_DOUBLE, neigh_proc, MyRank, MPI_COMM_WORLD);
/*.... Gather the result on the processor 0 ....*/
Output = (double *) malloc (n_size * sizeof(double));
MPI_Gather(X_buffer, NoofRows_Bloc, MPI_DOUBLE, Output, NoofRows_Bloc, MPI_DOUBLE, 0, MPI_COMM_WORLD);
/* .......Output vector .....*/
if (MyRank == 0) {
printf ("\n");
printf(" ------------------------------------------- \n");
printf("Results of Gaussian Elimination Method on processor %d: \n", MyRank);
printf ("\n");
printf("Matrix Input_A \n");
printf ("\n");
for (irow = 0; irow < n_size; irow++) {
for (icol = 0; icol < n_size; icol++)
printf("%.3lf ", Matrix_A[irow][icol]);
printf("\n");
}
printf ("\n");
printf("Matrix Input_B \n");
printf("\n");
for (irow = 0; irow < n_size; irow++) {
printf("%.3lf\n", Input_B[irow]);
}
printf ("\n");
printf("Solution vector \n");
printf ("\n");
for(irow = 0; irow < n_size; irow++)
printf("%.3lf\n",Output[irow]);
printf(" --------------------------------------------------- \n");
}
MPI_Finalize();
}
O segundo, tá nesse link http://technolog-world.blogspot.com.br/2010/02/mpi-program-that-implements-gauss_07.html
Provavelmente vou usar o segundo mesmo, pois ele gera dois arquivos de saída e também tá melhor organizado IMHO.
Parabéns pelo tutorial.
Estou com um problema. Olha se sabe qual o erro.
root@servidor:/usr/src/pvfs-2.8.2# /opt/pvfs2/bin/pvfs2-ping -m /mnt/pvfs2
/opt/pvfs2/bin/pvfs2-ping: error while loading shared libraries: libpvfs2.so: cannot open shared object file: No such file or directory
segui os passos e nao sei oque pode ser isso.
[33] Comentário enviado por ewerttonbd2 em 04/05/2014 - 16:39h:
Parabéns pelo tutorial.
Estou com um problema. Olha se sabe qual o erro.
root@servidor:/usr/src/pvfs-2.8.2# /opt/pvfs2/bin/pvfs2-ping -m /mnt/pvfs2
/opt/pvfs2/bin/pvfs2-ping: error while loading shared libraries: libpvfs2.so: cannot open shared object file: No such file or directory
segui os passos e nao sei oque pode ser isso.
/opt/pvfs2/bin/pvfs2-ping: error while loading shared libraries: libpvfs2.so: cannot open shared object file: No such file or directory
/opt/pvfs2/bin/pvfs2-ping: erro ao rodar a biblioteca compartilhada: libpvfs2.so: a biblioteca não pode ser aberta: não foi encontrado o arquivo ou diretório.
Se o sistema usado for o Debian do artigo execute:
# updatedb
# locate libpvfs2.so
e veja se a biblioteca existe. Se o comando não encontrar a biblioteca reveja os passos.
Bom dia!
Sabe me dizer se tem muita diferença no centos.
primeiramente obrigado pelo artigo, gostei muito
mas estou com um problema, quero fazer esse cluster semelhante ao seu, e estou com problema pra rodar programas proprietarios, o problema esta na hora de executar algum deles nas maquinas como user, fiz a instalação como su e dei todas as permissoes pro user poder executar o programa, mas mesmo assim diz que nao tenho permissoes. Esse mesmo procedimento funciona pra OpenSuse e Ubuntu, mas para Debian nao.
[35] Comentário enviado por willcamarg em 27/05/2014 - 13:04h:
Bom dia!
Sabe me dizer se tem muita diferença no centos.
Basicamente os comandos de instalação serão diferentes, mas a lógica é a mesma. Tu pode adaptar para o CentOs.
[36] Comentário enviado por henriqueuclides em 28/05/2014 - 14:51h:
primeiramente obrigado pelo artigo, gostei muito
mas estou com um problema, quero fazer esse cluster semelhante ao seu, e estou com problema pra rodar programas proprietarios, o problema esta na hora de executar algum deles nas maquinas como user, fiz a instalação como su e dei todas as permissoes pro user poder executar o programa, mas mesmo assim diz que nao tenho permissoes. Esse mesmo procedimento funciona pra OpenSuse e Ubuntu, mas para Debian nao.
Obrigado.
Mas não entendi tua dúvida.
Você está com dificuldades de instalar e rodar os programas proprietários?
Quais programas são esses?
Qual procedimento funciona para OpenSuse e Ubuntu e para Debian não ?
estamos instalando o GAUSSIAN09 e o MOLPRO em maquinas com Debian como super usuario, na instalação damos permissão para o usuario tambem poder executar esses programas, definimos o caminho no .profile do usuario, mas quando entramos como usuario diz que nao temos permissão.
Olá excelente Artigo Buckminster, eu tenho de criar um cluster mas com o ubuntu 12.04 você ou algum outro membro sabe como eu poderia fazer?
[39] Comentário enviado por henriqueuclides em 30/05/2014 - 13:41h:
estamos instalando o GAUSSIAN09 e o MOLPRO em maquinas com Debian como super usuario, na instalação damos permissão para o usuario tambem poder executar esses programas, definimos o caminho no .profile do usuario, mas quando entramos como usuario diz que nao temos permissão.
Provavelmente faltou alguma pasta à qual tu deve dar permissão.
Dê permissão recursivamente.
Tu instalou pelo apt-get ou compilou os programas?
[40] Comentário enviado por ricardo987 em 18/06/2014 - 19:50h:
Olá excelente Artigo Buckminster, eu tenho de criar um cluster mas com o ubuntu 12.04 você ou algum outro membro sabe como eu poderia fazer?
Obrigado.
No Ubuntu tu pode seguir basicamente o que que está no artigo, com algumas poucas adaptações.
Na página "Cluster 4", nesta parte:
Configurando a variável de ambiente, entrar no arquivo:
# vim ~/.bashcrc
o certo é
Configurando a variável de ambiente, entrar no arquivo:
# vim ~/.bashrc
Cara excelente artigo.
Atento ao tópicos e dicas.
Não executei mas você tem prazer em explicar, coisa rara hoje em dia.
Parabens!!
Na parte do pvfs2, esse erro aparece, alguem saber o motivo?
Testando:
# /opt/pvfs2/bin/pvfs2-ping -m /mnt/pvfs2
(3) Initializing each file system found in tab file: /etc/fstab...
PVFS2 servers: tcp://servidor:3334
Storage name: pvfs2-fs
Local mount point: /mnt/pvfs2
[E 12:03:13.439542] Error: BMI_sockio_connect_sock: (servidoor):: Unknown host
[E 12:03:13.439631] Send immediately failed: Unknown host
[E 12:03:13.439754] Warning: msgpair failed to tcp://no01:3334, will retry: Connection refused
[E 12:03:13.439774] Warning: msgpair failed to tcp://no02:3334, will retry: Connection refused
[E 12:03:13.439788] Warning: msgpair failed to tcp://no03:3334, will retry: Connection refused
[E 12:03:13.439802] Warning: msgpair failed to tcp://no04:3334, will retry: Connection refused
[E 12:03:13.439815] Warning: msgpair failed to tcp://servidoor:3334, will retry: Unknown host
/mnt/pvfs2: Ok
[44] Comentário enviado por thejack em 27/08/2014 - 08:52h:
Cara excelente artigo.
Atento ao tópicos e dicas.
Não executei mas você tem prazer em explicar, coisa rara hoje em dia.
Parabens!!
Obrigado.
[45] Comentário enviado por ewerttonbd2 em 09/09/2014 - 12:07h:
Na parte do pvfs2, esse erro aparece, alguem saber o motivo?
Testando:
# /opt/pvfs2/bin/pvfs2-ping -m /mnt/pvfs2
(3) Initializing each file system found in tab file: /etc/fstab...
PVFS2 servers: tcp://servidor:3334
Storage name: pvfs2-fs
Local mount point: /mnt/pvfs2
[E 12:03:13.439542] Error: BMI_sockio_connect_sock: (servidoor):: Unknown host
[E 12:03:13.439631] Send immediately failed: Unknown host
[E 12:03:13.439754] Warning: msgpair failed to tcp://no01:3334, will retry: Connection refused
[E 12:03:13.439774] Warning: msgpair failed to tcp://no02:3334, will retry: Connection refused
[E 12:03:13.439788] Warning: msgpair failed to tcp://no03:3334, will retry: Connection refused
[E 12:03:13.439802] Warning: msgpair failed to tcp://no04:3334, will retry: Connection refused
[E 12:03:13.439815] Warning: msgpair failed to tcp://servidoor:3334, will retry: Unknown host
/mnt/pvfs2: Ok
Provavelmente tu errou o nome de alguma máquina em algum arquivo (hosts, fstab, etc).
Veja a mensagem: Unknown host << host desconhecido.
Verifique os arquivos. Provavelmente é no servidor (tcp://servidoor:3334, will retry: Unknown host << tem dois 'os' na palavra servidor... é assim mesmo? Se for assim tenha o cuidado para estar igual em todos os arquivos).
Prezado
Tenho um cluster com suse server para processamento compartilhado, entretanto, para análise que precisa de memória ram não ocorre compartilhamento de mémoria ram, sendo usada somente a memória do nó de execução do programa (velvet). Existe como configurar para compartilhar memória?
Att,
Cícero
[48] Comentário enviado por cicerocarlos em 13/11/2014 - 01:32h:
Prezado
Tenho um cluster com suse server para processamento compartilhado, entretanto, para análise que precisa de memória ram não ocorre compartilhamento de mémoria ram, sendo usada somente a memória do nó de execução do programa (velvet). Existe como configurar para compartilhar memória?
Att,
Cícero
Qual é a biblioteca que tu utiliza? (Mpich, Openmpi, etc).
Estou usando a biblioteca mpich e mudei para o sistema ubuntu. O cluster fuciona muito bem, entretanto, não há compartilhamento de memória. Tenho que mudar a arquitetura do cluster ou posso configurar o mpich para compartilhar memória?
[50] Comentário enviado por cicerocarlos em 28/11/2014 - 01:17h:
Estou usando a biblioteca mpich e mudei para o sistema ubuntu. O cluster fuciona muito bem, entretanto, não há compartilhamento de memória. Tenho que mudar a arquitetura do cluster ou posso configurar o mpich para compartilhar memória?
Bom, tu configurastes a Mpich com o parâmetro --enable-shared ?
Veja a página Cluster 5 do artigo.
Acredito que no teu caso seja somente executar o ./configure com as opções que tu tinhas colocado anteriormente e acrescentar --enable-shared e depois executar make e make install.
Se não for isso então é algum outro problema.
Talvez alguma configuração no Velvet. Nunca trabalhei com este programa, mas no SIESTA, por exemplo, ele tem um gerenciamento próprio de memória.
Tu instalaste o Gânglia? Com ele tu podes ver se realmente a memória está sendo compartilhada.
De que forma tu detectaste que a memória não está sendo compartilhada... usaste algum programa para isso?
Eu instalei o mpich usando o apt-get do ubuntu e não criei as configurações do ./configure foram realizada pelo instalador. Eu descobri que não há compartilhamento de memória usando o top e monitorando o uso de memória, entretanto, o melhor parâmetro foi calcular e verificar que o velvet só analisa os reads (leituras de DNA) quando atinge o máximo de memória ram do nó. Após usar toda a memória, o processo paralisa o nó fica extremamente lento. Tem somo configura o --enable-shared? ou teria que construir um cluster na plataforma NUMA (pacote numactl)
Show de bola Buckminster, tá meio que de praxe elogiar, mas parabéns pelo artigo, rs.
[52] Comentário enviado por cicerocarlos em 28/11/2014 - 09:42h
Eu instalei o mpich usando o apt-get do ubuntu e não criei as configurações do ./configure foram realizada pelo instalador. Eu descobri que não há compartilhamento de memória usando o top e monitorando o uso de memória, entretanto, o melhor parâmetro foi calcular e verificar que o velvet só analisa os reads (leituras de DNA) quando atinge o máximo de memória ram do nó. Após usar toda a memória, o processo paralisa o nó fica extremamente lento. Tem somo configura o --enable-shared? ou teria que construir um cluster na plataforma NUMA (pacote numactl)
Desculpe a demora exagerada em responder.
Acredito que para configurar com o --enable-shraed tu terias que desinstalar através do apt-get e configurar e instalar o Mpich manualmente.
Quanto à plataforma NUMA (non-uniform memory access) nunca trabalhei com ela.
[53] Comentário enviado por bmarquesm em 07/02/2015 - 13:22h
Show de bola Buckminster, tá meio que de praxe elogiar, mas parabéns pelo artigo, rs.
Obrigado.
boa noite, não sei se entendia a definição de cluster direito , mas porque os back-end não são iguais nas instalações?
[56] Comentário enviado por linus black em 02/01/2016 - 23:08h
boa noite, não sei se entendia a definição de cluster direito , mas porque os back-end não são iguais nas instalações?
Tu está falando das máquinas nos nós (instalação física) ou está falando da instalação dos softwares?
Por favor me ajudem !
tentei dois jeitos
antes eu testei as chaves rsa
root@servidor: ssh 192.168.1.101
root@192.168.1.101's passoword:*****
root@no01~#: exit
OK !
ai quando executo :
root@servidor:/usr/src/mpich-3.0.4/examples# mpirun -hostfile /opt/hostfile -n 7 /usr/src/mpich-3.0.4/examples/cpi
Permission denied, please try again.
Permission denied, please try again.
Permission denied (publickey,password).
root@servidor:/usr/src/mpich-3.0.4/examples# mpirun 192.168.1.101 -n 7 /usr/src/mpich-3.0.4/examples/cpi
[proxy:0:0@servidor] HYDU_create_process (./utils/launch/launch.c:75): execvp error on file root@192.168.1.101 (No such file or directory)
===================================================================================
= BAD TERMINATION OF ONE OF YOUR APPLICATION PROCESSES
= EXIT CODE: 255
= CLEANING UP REMAINING PROCESSES
= YOU CAN IGNORE THE BELOW CLEANUP MESSAGES
===================================================================================
por favor URGENTE
[58] Comentário enviado por nguiovani em 09/10/2016 - 17:28h
Por favor me ajudem !
tentei dois jeitos
antes eu testei as chaves rsa
root@servidor: ssh 192.168.1.101
root@192.168.1.101's passoword:*****
root@no01~#: exit
OK !
ai quando executo :
root@servidor:/usr/src/mpich-3.0.4/examples# mpirun -hostfile /opt/hostfile -n 7 /usr/src/mpich-3.0.4/examples/cpi
Permission denied, please try again.
Permission denied, please try again.
Permission denied (publickey,password).
root@servidor:/usr/src/mpich-3.0.4/examples# mpirun 192.168.1.101 -n 7 /usr/src/mpich-3.0.4/examples/cpi
[proxy:0:0@servidor] HYDU_create_process (./utils/launch/launch.c:75): execvp error on file root@192.168.1.101 (No such file or directory)
===================================================================================
= BAD TERMINATION OF ONE OF YOUR APPLICATION PROCESSES
= EXIT CODE: 255
= CLEANING UP REMAINING PROCESSES
= YOU CAN IGNORE THE BELOW CLEANUP MESSAGES
===================================================================================
por favor URGENTE
Permission denied, please try again.
Permission denied, please try again.
Permission denied (publickey,password).
Faltou dar permissão a algum arquivo aí.
Olá Buckminster!
Parabéns pelo material, estou fazendo alguns testes, porém surgir esse erro abaixo, o arquivo está certo?
# vim /etc/bash.bashrc
# this file has to be sourced in /etc/profile.
if [ "$PS1" ] ; then
mkdir -m 0700 /dev/cgroup/cpu/user/$$
echo $$ > /dev/cgroup/cpu/user/$$/tasks
fi
Está apresentando o erro abaixo:
-bash: $'\302\240\302\240\302\240\302\240\302\240\302\240\302\240\302\240mkdir': comando não encontrado
-bash: /dev/cgroup/cpu/user/2395/tasks: Arquivo ou diretório não encontrado
[60] Comentário enviado por schileider em 02/12/2016 - 23:45h
Olá Buckminster!
Parabéns pelo material, estou fazendo alguns testes, porém surgir esse erro abaixo, o arquivo está certo?
# vim /etc/bash.bashrc
# this file has to be sourced in /etc/profile.
if [ "$PS1" ] ; then
mkdir -m 0700 /dev/cgroup/cpu/user/$$
echo $$ > /dev/cgroup/cpu/user/$$/tasks
fi
Está apresentando o erro abaixo:
-bash: $'\302\240\302\240\302\240\302\240\302\240\302\240\302\240\302\240mkdir': comando não encontrado
-bash: /dev/cgroup/cpu/user/2395/tasks: Arquivo ou diretório não encontrado
Provavelmente faltou a etapa anterior:
Criar o arquivo:
# vim /usr/local/sbin/cgroup_clean
Colocar o conteúdo:
#!/bin/sh
if [ "$1" != "/user" -a "$1" != "/user/" ]; then
rmdir /dev/cgroup/cpu$1
fi
Salvar e sair.
# chmod +x /usr/local/sbin/cgroup_clean
Patrocínio
Destaques
Artigos
Programa IRPF - Guia de Instalação e Resolução de alguns Problemas
Criando uma Infraestrutura para uma micro Empresa
Criar entrada (menuentry) ISO no Grub
Como gerar qualquer emoji ou símbolo unicode a partir do seu teclado
Dicas
Instalando o Pi-Hole versão v5.18.4 depois do lançamento da versão v6.0
Instalar o VIM 9.1 no Debian 12
Como saber o range de um IP público?
Muitas dificuldades ao instalar distro Linux em Notebook Sony Vaio PCG-6131L (VPCEA24FM)
Tópicos
Contas online no POP OS 24.04 ? (2)
problema para barrar sites/downloas via HTTPS. (2)